Hallo sobat100,
Sobat100, Hari rabu (03/10/2018) diumukannya peraih nobel 2018 di bidang kimia. Royal Swedish Academy of Sciences telah memutuskan siapa peraih Hadiah Nobel 2018 untuk Kimia. Sobat100, penasaran bukan siapa peraih nobel 2018 di bidang kimia ? Berikut peraih nobel di bidang kimia tahun ini . . .
Peraih Nobel Tahun 2018 Di bidang Kimia
Royal Swedish Academy of Sciences telah memutuskan untuk memberikan Hadiah Nobel untuk Kimia 2018 setengah bagian kepada Frances H Arnold "untuk mengatur perkembangan enzim", dan setengah bagian yang lain diberikan George P Smith dan Sir Gregory P Winter (masing-masing memperoleh seperempat bagian) "Phage untuk tampilan peptida dan antibodi â€.

(Frances H Arnold)

(George P Smith)

(Sir Gregory P Winter)
Latar Belakang
Perkembangan alami dari enzim telah ada sejak munculnya kehidupan di Bumi. Gen telah bermutasi dan protein telah berevolusi untuk meningkatkan kemampuan suatu organisme untuk mengatasi kondisi di lingkungan baru. Selama ribuan tahun, manusia telah membiakkan hewan dan tumbuhan melalui pemilihan organisme dengan sifat yang diinginkan. Untuk kebanyakan waktu ini mereka melakukannya tanpa mengetahui, manusia berevolusi dan mengoptimalkan enzim dan mengikat protein selama banyak generasi.

Gambar 1. Evolusi menuju evolusi terarah
Evolusi alami dari enzim telah ada sejak munculnya kehidupan di Bumi. Gen telah bermutasi dan protein telah berevolusi untuk meningkatkan kebugaran suatu organisme untuk mengatasi kondisi di lingkungan baru. Selama ribuan tahun, manusia telah membiakkan hewan dan tumbuhan melalui pemilihan organisme dengan sifat yang diinginkan. Untuk kebanyakan waktu ini mereka melakukannya tanpa mengetahui, manusia berevolusi dan mengoptimalkan enzim dan mengikat protein selama banyak generasi.
Evolusi enzim yang diarahkan dan protein yang mengikat telah menjadi strategi yang banyak digunakan di Indonesia penelitian akademis serta di industri kimia dan farmasi. Sutradara evolusi enzim-enzim menyesuaikan mereka untuk beroperasi dalam kondisi reaksi baru, mengoptimalkan aktivitas katalitik mereka terhadap substrat baru, dan membuat mereka mengkatalisasi reaksi kimia baru. Sutradara evolusi enzim telah memperluas repertoar biokatalis yang berguna. Enzim yang berevolusi menawarkan pilihan yang efisien dan ramah lingkungan untuk logam dan katalis organik dalam kimia dan industri bioteknik.
Evolusi enzim yang diarahkan dan protein yang mengikat - dalam teori
Pada tahun 1984, Manfred Eigen menerbitkan sebuah makalah teoritis yang menguraikan kemungkinan alur kerja untuk diarahkan evolusi enzim (Gambar 2, ref 1). Eigen mencatat bahwa pengoptimalan seperti itu menjadi tantangan yang menarik karena genotipe dan fenotipe bergantung pada molekul yang berbeda. Dia beralasan bahwa menemukan varian langka di perpustakaan besar, jika bukan langka akan sulit dan mustahil. Sebaliknya ia mengusulkan penggunaan perpustakaan yang lebih kecil dan beberapa generasi mutagenesis dan skrining sebagai prosedur yang kemungkinan besar akan mengarah ke depan. Eigen memprediksi bahwa tidak mungkin untuk membangun "mesin evolusioner" bertahap yang berulang-ulang untuk menghasilkan enzim yang optimalkan.

Gambar 2. Kutipan dari proposal teoritis Eigen tentang evolusi terarah.(1)
Prediksi teoritis lain ditemukan dalam hak cipta (2) yang menggambarkan evolusi protein yang mengikat melalui diversifikasi perpustakaan yang berulang antara putaran seleksi. Evolusi enzim yang disutradarai juga diperkenalkan secara singkat sebagai kemungkinan dalam sebuah ringkasan dari uji protein-engineering yang lebih klasik dari optimasi enzim.(3)
Evolusi enzim yang diarahkan pada praktiknya
Satu dekade setelah karya teoritis Eigen,(1) karya eksperimental pertama muncul yang menggambarkan keberhasilan penerapan langsung evolusi enzim dalam pengaturan laboratorium untuk meningkatkan fungsi enzim dan fleksibilitas.(4) Frances H Arnold melaporkan evolusi yang diarahkan dari subtilisin E untuk mendapatkan varian enzim yang aktif dalam lingkungan yang sangat tidak alami (denaturasi), yaitu pada konsentrasi tinggi pelarut polar organik dimetilformamida (DMF). Setelah empat putaran berurutan dari mutagenesis dan skrining di Kehadiran DMF, varian enzim dengan aktivitas 256 kali lipat lebih tinggi daripada enzim tipe liar di 60% (v / v) diciptakan DMF.(4)(5)
Dalam makalah seminal,(4) Arnold telah menguasai seluruh alur kerja untuk evolusi enzim terarah, metodologi yang mengandalkan beberapa bagian: 1) identifikasi enzim awal yang sesuai untuk tugas yang dipilih, 2) pembuatan pustaka urutan DNA untuk mencakup sub bagian yang dipilih dengan baik ruang sekuens, 3) seleksi identifikasi kriteria yang akan mengarah pada peningkatan atau fungsi baru dan metode pemilihan varian enzim yang dioptimalkan, 4) diversifikasi kembali oleh gen ke membuat pustaka urutan DNA baru di sekitar urutan dari pilihan pertama untuk menutupi himpunan yang baru dari bagian ruang sekuens, 5) pengaturan pemilihan kriteria dengan meningkatkan kekakuan, dan untuk seterusnya sebanyak putaran yang diperlukan untuk mencapai tingkat target kinerja enzim. Masing-masing dari lima ini langkah-langkah telah dikembangkan dan dioptimalkan lebih lanjut selama bertahun-tahun di labolatorium Arnold dan beberapa laboratorium lainnya.
Selain sau set pertama dari empat mutasi tunggal gabungan, pekerjaan pertama(4) menggunakan kesalahan-rawan PCR untuk membuat dan melakukan diversifikasi ulang pustaka urutan-DNA melalui tiga putaran acak mutagenesis dan skrining untuk berevolusi subtilisin E. Kriteria seleksi adalah hidrolisis dari kasein protein susu. Varian enzim aktif menciptakan lingkaran cahaya yang terlihat pada piring agar dengan kasein. Enzim yang disekresikan oleh koloni bakteri dengan demikian dipindahkan ke pelat agar yang mengandung keduanya DMF dan kasein, untuk memungkinkan identifikasi varian enzim yang paling aktif di hadapan pelarut organik. DNA plasmid diisolasi dari klon yang mensekresikan varian enzim itu menghasilkan halo yang lebih besar daripada yang mengelilingi induk enzim, dan mengalami lebih lanjut putaran mutagenesis.
Evolusi yang diarahkan dari subtilisin E untuk meningkatkan aktivitas dalam pelarut organik polar adalah patokan pencapaian yang membuka bidang evolusi enzim yang diarahkan. Pekerjaan ini menjadi titik awal untuk pengembangan teknis lanjutan dari metodologi untuk diarahkan evolusi. Medan diperluas untuk memperbaiki dan membentuk kembali enzim untuk banyak reaksi kimia, lama dan baru, yang mengarah ke aplikasi penting untuk penelitian organik sintesis, serta untuk industri kimia dan farmasi dan seterusnya.
Panduan pembuatan wawasan perpustakaan molekuler
Tidak mungkin untuk mengacak setiap posisi dalam suatu enzim, ukuran umumnya adalah 200-300 residu asam amino atau lebih. Memang, hanya sebagian kecil dari posisi asam amino yang bisa bervariasi jika tujuannya adalah perpustakaan dengan cakupan urutan penuh. Alasannya sederhana kombinatorial matematika dan jumlah varian yang berkembang pesat relatif terhadap jumlah klon itu dapat ditangani di pengaturan laboratorium apa pun, atau bahkan menggunakan kapasitas gabungan semua laboratorium di Indonesia Dunia. Namun, banyak sekali penelitian yang menjelaskan bahwa mutasi di dan di dekat situs aktif sebagai serta substitusi yang lebih jauh pada permukaan enzim dapat berkontribusi pada katalitik yang dioptimalkan aktivitas. Arnold dan rekan kerja telah ditunjukkan oleh banyak contoh bahwa desain perpustakaan harus berdasarkan wawasan molekuler dan pilihan berbasis pengetahuan di mana posisi asam amino bervariasi, dikombinasikan dengan beberapa elemen keacakan tambahan, misalkan melalui PCR yang rawan kesalahan.
Kontributor awal yang menonjol untuk pengembangan dan implementasi metodologi untuk evolusi terarah adalah almarhum William (Pim) Stemmer († 2013). Stemmer memperkenalkan strategi rekombinasi DNA yang disebut "DNA shuffling" ke evolusi enzim. Ini adalah cara yang efisien untuk menyebarkan mutasi yang bermanfaat sambil meningkatkan ukuran pustaka DNA melalui fragmentasi acak dan merakit kembali gen.(6)(7)Dia menunjukkan bahwa penggunaan DNA pengacakan, yaitu rekombinasi DNA dari gen serupa dari beberapa organisme, introduksi lebih banyak variasi daripada banyak metode lain dan dengan demikian dapat meningkatkan peluang untuk mencapai peningkatan aktivitas substansial dalam varian yang berevolusi. Dalam studi bukti-prinsip Stemmer dan rekan kerja berangkat untuk meningkatkan aktivitas enzim -laktamase (enzim yang bertanggung jawab untuk resistensi antibiotik); tiga siklus mengocok DNA dan skrining pada piring dengan berturut-turut konsentrasi yang lebih tinggi dari cefotaxime antibiotik menyebabkan evolusi enzim dengan signifikan peningkatan aktivitas.(6)(7) Pengacakan gen telah dilaporkan sebagai sarana untuk meningkatkan variasi dan meningkatkan afinitas antibodi untuk target, kadang-kadang disebut pematangan afinitas, dan diberikan lebih awal contoh evolusi terarah dari protein pengikat.(8-10)
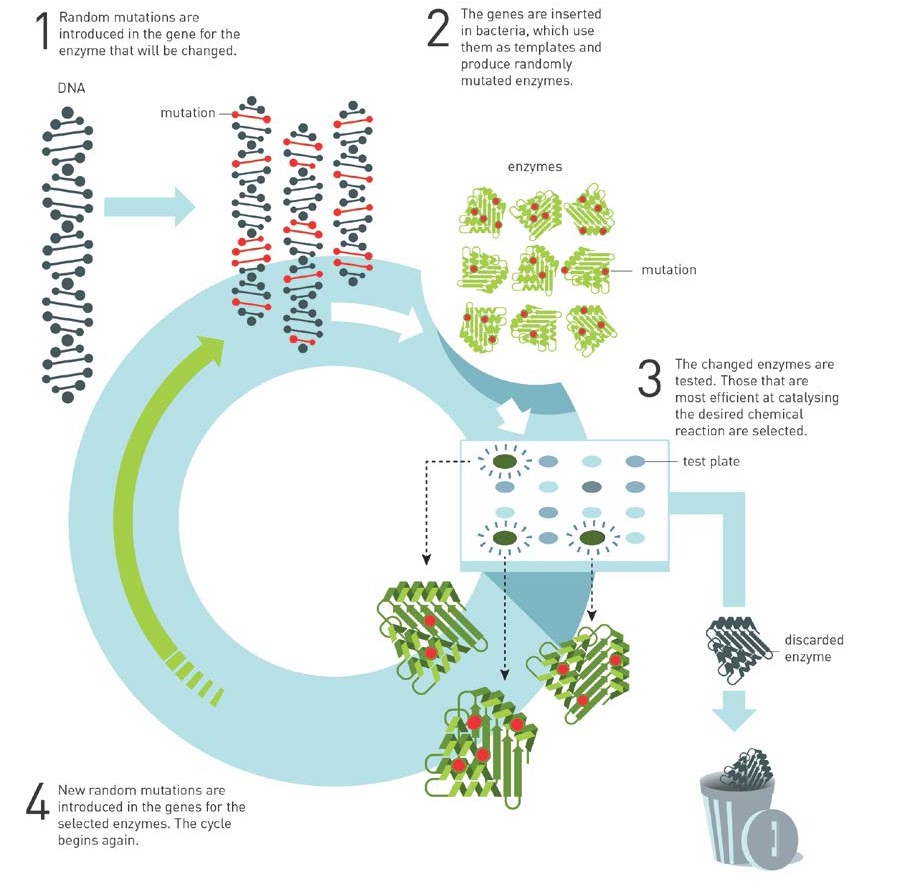
Gambar 3. Aliran kerja untuk evolusi enzim yang diarahkan.
Dalam kasus ini gen yang dikocok segmen berhubungan dengan rantai ringan (10) atau ke loop variabel rantai berat dan ringan imunoglobulin (8)(9)DNA shuffling, proses staggered extension(StEP) dan metode lain untuk generasi perpustakaan yang kembangkan lebih lanjut di laboratorium Arnold dan Stemmer untuk digunakan dalam evolusi yang diarahkan enzim selama paruh kedua tahun 1990-an.(11-26) Sejak akhir abad lalu, secara progresif menurunkan biaya untuk sintesis gen de novo dengan kodon yang merosot, atau sepenuhnya mendesain perpustakaan DNA, telah membuka jalan baru menuju produksi yang efisien dan terjangkau urutan perpustakaan dengan keragaman yang disesuaikan.
Kriteria seleksi dan teknik skrining
Kriteria seleksi dan teknik skrining harus disesuaikan dengan setiap optimasi enzim berusaha keras. Seleksi dapat digabungkan dengan fungsi kelangsungan hidup sel; misalnya, Aktivitasyang diinginkan enzimatik dapat mendetoksifikasi senyawa yang sebaliknya menghambat pertumbuhan. Seleksi mungkin juga digabungkan ke uji enzim spektroskopi atau cara lain dari probe optik atau okular untuk meningkatan aktivitas.
Ketika menggunakan evolusi diarahkan untuk meningkatkan aktivitas enzim dalam kondisi tidak asli, seperti suhu tinggi atau konsentrasi tinggi zat beracun atau denaturasi atau pelarut organik, kondisi mungkin harus diperkenalkan bertahap dengan tingkat ketajaman yang meningkat secara bertahap tekanan seleksi. Ini memastikan bahwa contoh enzim di setiap putaran memiliki setidaknya beberapa Aktivitas awal yang belum sempurna di bawah kondisi yang digunakan dalam putaran itu. Peningkatan bertahap dari faktor tekanan, dengan intervensi diversifikasi antara putaran seleksi, memungkinkan untuk memperoleh enzim atau enzim yang lebih efektif dan lebih toleran dengan properti katalitik baru. Seleksi dapat dilakukan pada piring agar, mengangkat menggunakan uji filter atau menggunakan flow cytometry.(27)(28) Dan Tawfik menunjukkan bahwa evolusi enzim yang terarah dapat dibentuk tanpa menggunakan sel hidup, misalnya, menggunakan kompartementalisasi in-vitro dalam air dalam minyak tetesan emulsi mengandung ribosom dan perpustakaan mRNA .(29)
Kondisi reaksi baru
Aplikasi awal evolusi enzim diarahkan bertujuan untuk mengoptimalkan stabilitas dan kinerja di bawah kondisi reaksi baru seperti tinggi fraksi pelarut organik (4), putaran lebih lanjut dari evolusi diarahkan ditambahkan untuk mencapai peningkatan aktivitas 471 kali lipat di atas wildtype.(30) Contoh lain dari labolatorium Arnold menyangkut pengoptimalan paranitrobenzil esterase untuk aktivitas di hadapan.(31)
Banyak metode yang ada untuk meningkatkan termostabilitas enzim dan protein lainnya. Ketika evolusi diarahkan enzim digunakan dengan tujuan untuk meningkatkan stabilitas termal mereka, yang proses evolusi dapat diatur sebagai perlakuan panas yang disisipkan dan tes aktivitas, atau tes aktivitas apat dilakukan pada suhu tinggi. Dalam satu uji, termostabilitas Bacillus subtilis p-nitrobenzyl esterase meningkat lebih dari 14 ° C (meningkat di Tm) setelah enam generasi mutagenesis acak, rekombinasi melalui DNA shuffling, dan skrining dengan perlakuan panas interleaved dan tes aktivitas.(32) Pekerjaan ini menunjukkan itu mungkin untuk meningkatkan stabilitas termal enzim tanpa mengorbankan katalitiknya aktivitas pada suhu yang lebih rendah, jika kedua properti dibatasi. Jika tidak, evolusi satu properti mungkin datang dengan biaya yang lain, terlepas dari apakah kedua properti itu berkorelasi terbalik atau tidak berkorelasi sama sekali.(32) Alam biasanya menyediakan organisme yang disesuaikan lingkungan dingin atau hangat dengan dua enzim yang berbeda memiliki sifat katalitik yang optimal rendah atau pada suhu tinggi, masing-masing. Arnold menunjukkan bahwa evolusi terarah dapat menghasilkan enzim tunggal dengan aktivitas katalitik tinggi pada suhu tinggi dan rendah.(32-35) Lain strategi evolusi diarahkan bergantung pada informasi struktural dalam bentuk Bfactors kristalografi, ukuran wilayah mana yang kurang lebih dipesan dalam protein yang dikristalkan. Dengan memfokuskan pustaka mutasi ke 10 posisi dengan B-factor tertinggi, peningkatan besar stabilitas enzim tercapai.(36)
Selain untuk mendapatkan biokatalis baru yang lebih baik, penelitian evolusi yang diarahkan berkontribusi pemahaman umum kita tentang proses evolusi protein alami dan determinan enzim tindakan, meskipun tekanan seleksi beroperasi pada skala waktu yang sangat berbeda, populasi ukuran, tingkat mutasi, kekuatan seleksi, dll. Arnold dan yang lain telah menunjukkan pentingnya untuk evolusi protein faktor seperti termostabilitas,(37)(38) efek relatif dari acak mutasi dan rekombinasi,(39) pentingnya pergeseran netral untuk evolusi protein fungsi (40)(41) dan korelasi antara tingkat ekspresi protein dan evolusi.(42)
Pilihan Negara Awal
Arnold dan rekan kerja telah berulang kali menunjukkan bahwa mungkin untuk mengembangkan enzim untuk memperbaiki aktivitas mereka di bawah kondisi baru dalam hal komposisi solusi, suhu, dll, dan untuk mengubah aktivitas katalitik mereka menjadi substrat dan reaksi baru. Ini mungkin selama enzim yang dipilih sebagai titik awal memiliki setidaknya beberapa tingkat aktivitas rendah untuk reaksi yang dimaksudkan, yaitu beberapa tingkat pergaulan katalitik (Gambar 4, ditinjau dalam misalnya 43-46). Sebuah perancah tidak aktif bukanlah pilihan yang cocok; evolusi terarah memerlukan beberapa tingkat aktivitas rendah. Bahkan tingkat aktivitas yang sangat rendah terhadap reaksi yang dimaksudkan memberikan keadaan awal untuk mengoptimalkan melalui evolusi. Seringkali hanya beberapa mutasi yang diperlukan untuk meningkatkan aktivitas baru.
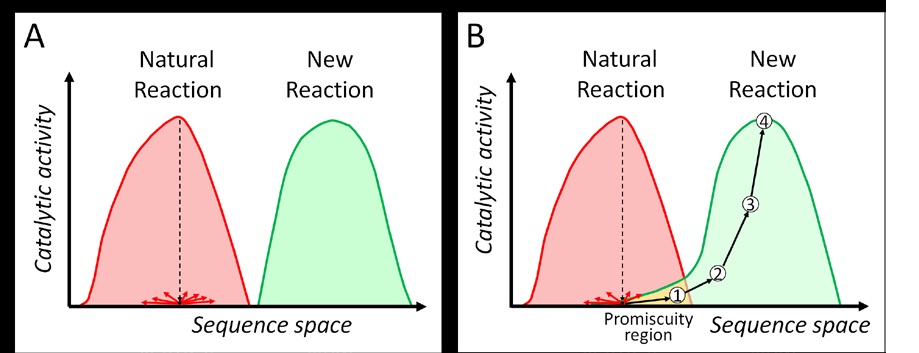
Gambar 4. A: Titik awal tanpa aktivitas untuk reaksi yang dimaksud tidak berguna karena tidak ada variasi urutan (panah merah) menciptakan reaktivitas baru. B: Enzim promiscuous dengan setidaknya aktivitas rendah untuk reaksi yang dimaksudkan adalah titik menatap yang cocok. Beberapa kombinasi mutasi acak dapat meningkatkan reaktivitas baru (panah hitam). Varian pertama (1) berfungsi sebagai keadaan awal untuk putaran berurutan variasi dan penyaringan (2) (3) (4) untuk varian yang lebih baik. Hanya sejumlah kecil siklus dan biasanya diperlukan untuk menaikkan reaktivitas baru.
Jika suatu enzim memiliki tingkat aktivitas yang rendah untuk reaksi yang diinginkan, tetapi aktivitas yang lebih tinggi untuk yang alami, mungkin bermanfaat untuk lebih dulu menurunkan aktivitas alami sebelum memulai upaya evolusi diarahkan menuju reaksi yang dimaksudkan.
Reaksi kimia baru
Sebagai contoh terbaru dari strategi terakhir ini, aktivitas triptofan sintetase dari Pyrococcus furiosus pertama kali dikurangi oleh 95% melalui penghapusan domain non-katalitik dari enzim. Domain katalitik yang terisolasi tunduk pada tiga putaran evolusi diarahkan ke memperkenalkan kegiatan katalitik baru menuju sintesis analog tryptophan.(47-51)
Dalam serangkaian penelitian, Arnold dan rekan kerja mengubah aktivitas sitokrom P450 menjadi katalisasi serangkaian reaksi yang tidak ada spesifik enzim yang sebelumnya tersedia, misalnya, siklopropanasi. Sitokrom P450BM3 memiliki pergaulan katalitik dan kemampuan untuk mengkatalisasi, dengan efisiensi yang sangat rendah, siklopropanasi styrene oleh etil-diazoasetat (EDA). Banyak enzim yang lebih spesifik dan efisien berevolusi dan hanya sebagian kecil (0,2%) dari amino Asam dalam enzim perlu diubah untuk mengoptimalkan aktivitas katalitik baru.(52-54) Ini termasuk perubahan dari residu ligasi besi dari Cys ke Ser atau-Nya, yang mengarah ke pergeseran dalam karakteristik 450-nm puncak Soret dalam spektrum absorbansi enzim ke 411 nm. Oleh karena itu, enzim yang berevolusi disebut sitokrom P411.
Contoh lain dari reaksi yang tidak ada enzim alami telah berevolusi adalah transfer nitrene reaksi. Dalam satu kasus, Arnold dan rekan kerja mulai dari varian sitokrom P411 berkinerja reduksi azide sekitar 100 kali lebih efisien daripada transfer nitrene ke sulfida. Menggunakan mengarahkan evolusi mereka menghasilkan varian enzim yang justru mempromosikan secara efisien proses transfer nitrene yang diinginkan.(55) Ada beberapa contoh lain dari evolusi yang diarahkan enzim untuk reaksi transfer karbene dan nitrene (lihat misalnya 56,57).
Reaksi dengan ikatan CH alifatik dan aromatik adalah tujuan lain yang bisa dikerjakan. Menggunakan diarahkan evolusi cytochrome P450 monooxygenase, enzim yang dibuat yang mengkatalisis aminasi intermolekular dari ikatan C-Hm benzilik. Biokatalis bersifat enansioselektif dan bertahan lama hingga 1.300 turnovers, sehingga memberikan biokatalis yang efisien untuk sintesis berharga amina benzilik.(58)
Contoh lain dari evolusi terhadap reaksi baru termasuk pembentukan enzim yang mengkatalisasi detoksifikasi arsenat ,(14) produksi karbocycles yang sangat tegang,(59) dan beralih dari enzim dari galactosidase ke fucosidase.(15)
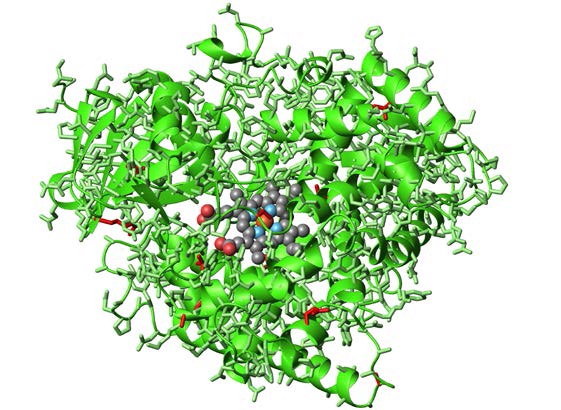
Gambar 5. Biokatalis yang berevolusi untuk siklopropanasi. Varian sitokrom P411 dari sitokrom P450 (ref. 52) dengan tulang punggung protein ditampilkan sebagai representasi pita dan rantai samping sebagai tongkat. Sisi-rantai yang bermutasi dalam varian rekayasa ditampilkan dalam warna merah.
Jalur metabolik
Kekuatan dari metodologi evolusi terarah adalah kemampuan untuk berevousi bersama enzim di jalur biosintesis. Dalam satu contoh, Arnold dan rekan kerja mengembangkan jalur multi-enzim untuk produksi karotenoid dalam E. coli.(60) Laboratoriumnya juga menunjukkan bagaimana biokatalis sel utuh bisa dikembangkan untuk produksi bahan kimia berharga dengan menggunakan evolusi yang diarahkan untuk mengaktifkan produksi L-metionin dalam E. coli.(61)
Biofuels
Satu tantangan bagi umat manusia adalah menemukan pengganti atau suplemen yang sesuai untuk bahan bakar fosil, yang dapat diproduksi dengan cara yang ramah lingkungan dan berkelanjutan. Di sini, seseorang mencari untuk menghasilkan alkohol dari alkana rantai pendek (62) dan kandidat utama biofuel adalah 2- methylpropan-1-ol (isobutanol). Isobutanol dapat diproduksi menggunakan jalur biosintetik di Escherichia coli rekombinan. Bagaimanapun enzim di dua jalur, membutuhkan pengurangan nicotinamide adenine dinucleotide phosphate (NADPH) sebagai kofaktor, sementara glikolisis, metabolisme normal selama pertumbuhan E. coli menghasilkan penurunan adenin nikotinamida dinukleotida (NADH). Untuk mengatasi hambatan ini, Arnold dan rekan kerja menggunakan evolusi terarah untuk mengubah ketergantungan co-factor dari enzim sehingga mereka dapat bergantung pada NADH, membuat enzim dan dengan demikian organisme cocok untuk produksi biofuel.(63)
Ikatan kimia baru
Ikatan karbon-silikon umum dalam bahan kimia buatan manusia tetapi tidak ada dalam biologi. Alam memiliki tidak berevolusi enzim yang mengkatalisis pembentukan ikatan karbon-silikon. Namun, diarahkan evolusi dapat digunakan sebagai strategi untuk memastikan bahwa kimia semacam itu yang ditemukan oleh manusia juga bisa dilakukan dengan bantuan enzim. Arnold dan rekan kerja mencatat bahwa protein haem dapat mengkatalisasi reaksi penyisipan carbene non-alami. Setelah skrining sejumlah protein haem dari berbagai organisme, mereka memutuskan untuk menggunakan sitokrom c dari Rhodothermus marinus sebagai permulaan titik. Protein ini mengkatalisis pembentukan ikatan silikon-karbon dengan efisiensi rendah, tetapi dengan 97% kelebihan enansiomerik.(64) Sebuah perpustakaan kecil varian disaring selama panas pengobatan dan dalam uji aktivitas katalitik, dan kandidat terbaik menjadi sasaran lebih lanjut mutagenesis dan skrining. Hasil dari pekerjaan ini adalah enzim yang mengkatalisis silikon-karbon pembentukan ikatan 40 kali lebih baik daripada enzim awal dan dengan 99% ee .(64)Yang berevolusi enzim memiliki jumlah perputaran 15 kali lebih tinggi daripada katalis non-enzim terbaik yang dikenal untuk reaksi yang sama.
Contoh ini menunjukkan bahwa adalah mungkin untuk memperluas ruang lingkup katalis enzim reaksi dalam hal jenis ikatan yang dibentuk oleh enzim rekayasa. Contoh lain dari ikatan dan reaksi tidak dikatalisis oleh enzim apa pun yang ditemukan di alam, tetapi untuk yang mengarahkan evolusi digunakan untuk membuat enzim yang efisien, adalah ikatan karbon-borongan (65) dan amina C-H intramolekuler enantio-selektif .(66)
Selektivitas Enantio
Sutradara evolusi adalah cara yang efisien untuk meningkatkan enantio-selektivitas enzim, yaitu, meningkatkan kinerja mereka dalam katalisis asimetris. Enzim yang berevolusi digunakan dalam produksi zat kiral dengan kemurnian enansiomer tinggi. Contoh awal diarahkan evolusi dengan tujuan meningkatkan enantio-selektivitas enzim dilaporkan oleh Matcham dan Bowen mengenai transaminase dalam katalisis produksi amina kiral.(67) Pekerjaan ini dimulai dengan enzim dengan tingkat selektivitas S rendah (65% ee) dalam konversi keton -tetralone menjadi aminotetraline, amina yang sesuai. Perpustakaan mutan adalah dihasilkan dan disaring untuk peningkatan aktivitas pada S-isomer tetapi bukan R-isomer. Hasil adalah biokatalis yang menghasilkan S-aminotetraline dengan selektivitas yang sangat ditingkatkan (94% ee), yang lebih ditingkatkan dengan putaran tambahan mutagenesis dan skrining.(67) Manfred Reetz dan rekan kerja melaporkan contoh awal lain yang telah menyebabkan peningkatan enantioselektivitas lipase dalam hidrolisis ester.(68) Melalui evolusi yang diarahkan melalui empat siklus mutagenesis acak, faktor selektivitas lipase bakteri dari Pseudomonas aeruginosa pertama kali ditingkatkan dari 1,1 menjadi 11 (68) dan kemudian menjadi 35 setelah diversifikasi lebih lanjut dari perpustakaan.(69) Contoh awal lainnya ditemukan.(70-73)
Sutradara evolusi dalam sintesis organik dan industri
Evolusi yang disutradarai dengan cepat berjalan dari pengaturan akademik ke aplikasi industri.(74-77) Enzim yang dikembangkan menggunakan evolusi diarahkan digunakan dalam industri dalam produksi biofuel, bahan, curah dan bahan kimia, deterjen, produk konsumen, reagen laboratorium dan obat-obatan, serta perantara untuk industri farmasi. Beberapa dari enzim yang dikembangkan di lab Arnold digunakan dalam industri. Banyak perusahaan memiliki milik mereka sendiri tim ilmiah menerapkan strategi evolusi terarah untuk meningkatkan katalis atau berbasis protein terapeutik dalam hal stabilitas, aktivitas, spesifisitas atau properti lainnya. Contoh spesifik dari berevolusi enzim dan produk peningkat rasa, obat-obatan melawan diabetes dan plak pembuluh darah, serta obat-obatan penurun lipid. Beberapa enzim yang diproduksi oleh evolusi diarahkan dibuat dalam skala sangat besar. Ini termasuk lipase yang digunakan dalam deterjen. Bahan kimia industri dibuat dalam jumlah besar dengan bantuan biokatalis yang diproduksi oleh evolusi yang diarahkan.
Sutradara evolusi
Pada desain protein Evolusi yang diarahkan berusaha mengubah aktivitas enzim yang sudah ada berdasarkan molekuler wawasan dikombinasikan dengan unsur besar keacakan. Orthogonal untuk mengarahkan evolusi adalah desain protein yang rasional. Desain protein didasarkan pada ab initio atau perhitungan empiris dan bertujuan untuk mendesain protein dari awal. Namun, di bidang desain protein, itu sangat luas mengakui bahwa untuk mencapai tingkat kebugaran yang dapat diterima, misalnya, dalam hal mengikat afinitas dan spesifisitas, itu pada tingkat pengetahuan kami saat ini yang diperlukan untuk menambahkan diarahkan evolusi sebagai langkah optimasi akhir (lihat misalnya referensi. 78-80).
Tampilan phage dari peptida
Dalam makalah seminal, Smith menunjukkan bahwa peptida dapat dimasukkan ke dalam lingkaran protein III, a minor coat protein pada permukaan fag fusi, dan peptida yang ditampilkan dipertahankan interaksi dengan targetnya.(81) Dalam karya ini, peptida yang ditampilkan adalah 57-residu fragmen sebuah endonuklease restriksi. Menggunakan satu putaran pemurnian afinitas versus antiserumke endonuklease, Smith menunjukkan bahwa fag menyajikan peptida endonuklease yang dimasukkan ke dalam mantel Protein III dari permukaannya dapat diperkaya 1.000 kali lipat dibandingkan fag lain. Dia menguraikan cara maju menuju pengayaan yang jauh lebih besar, yang mungkin dicapai dengan menggunakan urutan putaran pemurnian afinitas. Dia juga meramalkan bahwa mungkin untuk mengisolasi klon dari a perpustakaan sisipan acak dalam vektor fag menggunakan pemurnian afinitas dengan antibodi sebagai umpan.
Dalam publikasi lain, dari 1988,(82) Parmley dan Smith memperkenalkan beberapa teknik perbaikan pada teknologi tampilan phage, misalnya, dengan memindahkan lokasi peptida yang ditampilkan dalam protein III. Ini dimotivasi oleh fakta bahwa protein III menengahi infeksi E. coli dengan mengikat ke F pilusnya. Protein III yang berfungsi penuh dengan demikian penting untuk propagasi dari fag. Smith memperkenalkan istilah "biopanning" untuk pemurnian afinitas phages menampilkan peptit afinitas tinggi dari latar belakang afinitas yang lebih rendah, menggunakan Piring Petri berlapis streptavidin yang menjadi target biotinilasi digabungkan. Ini menawarkan hal yang mudah perangkat untuk menangkap, mencuci dan elusi dalam prosedur pemurnian afinitas. Smith menunjukkan kelayakan pendekatannya ditingkatkan dengan mencapai 108 kali lipat pengayaan phages menampilkan a peptida mewakili epitop antibodi anti--galactosidase menggunakan pemurnian afinitas menuju targetnya. Smith juga mengusulkan agar tampilan peptida dapat membantu dalam pengembangan vaksin. Ini studi terilhami di mana peptida dari parasit malaria Plasmodium falciparum adalah ditampilkan pada permukaan phage berfilamen dan ditemukan aktif sebagai antigen.(83)(84)
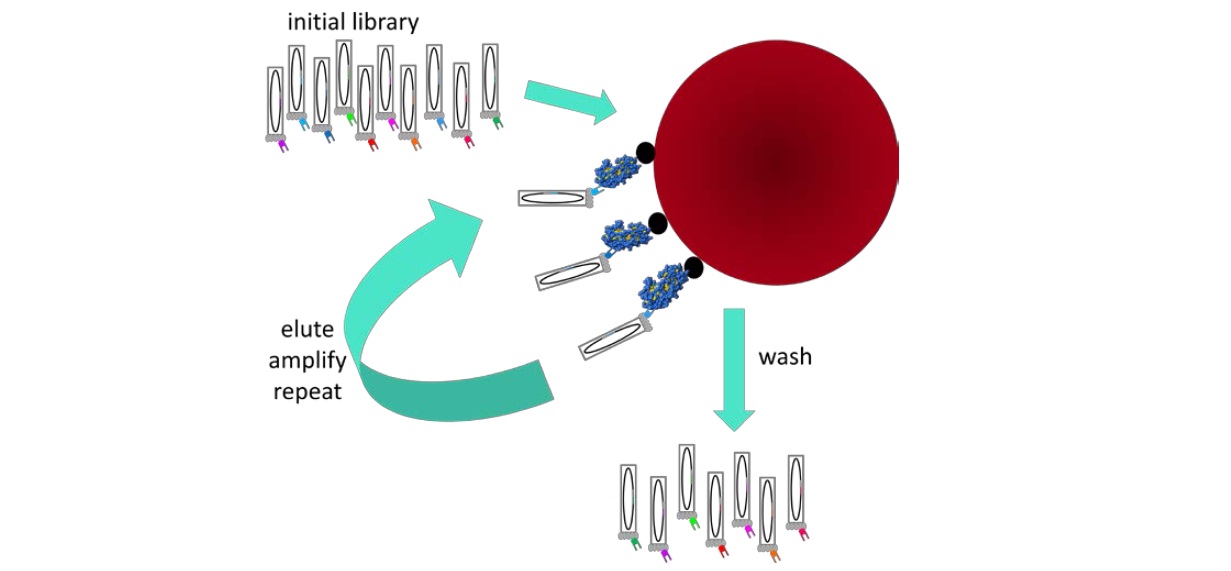
Gambar 6. Pemilihan protein pengikat afinitas tinggi dari perpustakaan tampilan phage. Pustaka awal peptida atau varian protein ditampilkan pada protein III di ujung phage (kiri atas) ditambahkan ke protein target diimobilisasi pada dukungan yang solid, di sini manik-manik magnetik (merah). Setelah mencuci luas untuk menghapusnya dengan lemah phage yang terikat (kanan bawah), varian pengikatan terbaik dielusi (misalnya menggunakan asam) dan digunakan untuk menginfeksi E coli ke menghasilkan pengayaan perpustakaan yang diperkuat pada anggota afinitas tinggi untuk putaran berikutnya pemurnian afinitas. Itu prosedur dapat diulang sebanyak yang diperlukan untuk mendapatkan tingkat seleksi yang diinginkan.
Tampilan phage peptida perpustakaan
Dua makalah pertama muncul dalam edisi Science yang sama.(85)(86) Kedua perpustakaan itu diekspresikan dalam fusi dengan coat protein III. Devlin dan rekan kerja melaporkan perpustakaan 20 juta peptida 15-mer yang berbeda, yang digunakan untuk menyimpulkan motif pengikatan peptida, HPQ, yang menganugerahkan afinitas tinggi untuk situs pengikatan biotin streptavidin.(85)
Scott dan Smith melaporkan perpustakaan 40 juta peptida 6-mer yang digunakan untuk menentukan epitop dari dua antibodi monoklonal berbeda yang telah dibangkitkan terhadap fragmen enam residu myohaemerythrin.(86) Peptida yang mengikat antibodi dipasangi afinitas dari perpustakaan melalui tiga putaran biopanning berturut-turut, diikuti oleh infeksi E. coli untuk menghasilkan pustaka display phage yang diperkaya untuk putaran berikutnya, dan seterusnya. Setelah tiga putaran afinitas pemurnian, dikombinasikan dengan pengukuran afinitas untuk klon yang dipilih, Smith dan rekan kerja menunjukkan bahwa hanya peptida afinitas yang sangat tinggi yang dipertahankan. Motif konsensus yang mengikat untuk epitop antibodi dapat disimpulkan berdasarkan sekuensing DNA klon individu.(86) Oleh membandingkan sejumlah besar sekuens asam amino, mereka menemukan bahwa tiga asam amino pertama dari epitop yang paling penting untuk berunding afinitas tinggi yang mengikat pada antibodi.
Perpustakaan acak hexa-peptida digunakan untuk memilih peptida pengikat afinitas tinggi untuk anti-endorphin antibodi; setelah tiga putaran pemurnian afinitas dan sekuensing 51 klon, a motif konsensus dapat disimpulkan dan dua residu N-terminal yang paling ditemukan menjadi paling penting dalam epitop ini untuk menentukan afinitas antibodi.(87) Tampilan phage peptida perpustakaan memungkinkan untuk menentukan epitop untuk antibodi. Melalui tampilan fag dari antibodi, peneliti dapat membalikkan prosedur seleksi,yang merupakan langkah penting menuju pengembangan antibodi terapeutik.
Tampilan phage dari fragmen antibodi
Pada tahun 1990, Sir Gregory P Winter melaporkan tampilan antibodi terlipat dan berfungsi penuh fragmen pada phage berfilamen.(88) IgG utuh adalah molekul bivalen besar yang terdiri dari lebih banyak dari satu rantai asam amino dan oleh karena itu menantang untuk mengekspresikan pada permukaan Phage. Musim dingin dan rekan kerja malah menampilkan fragmen variabel berantai tunggal, scFv, sebuah konstruksi di mana bagian variabel dari rantai berat secara kovalen terhubung melalui linker polipeptida fleksibel ke rantai ringan.(89) Dengan demikian, scFv membawa situs pengikatan antigen tunggal yang terdiri dari enam variabel loop, yang disebut daerah yang saling melengkapi (CDRs).
Dalam karya pertama, Musim Dingin dan rekan kerja menunjukkan scFv yang diturunkan pada tikus yang diimunisasi terhadap lisozim telur-putih dalam fusi dengan protein phage filamen III. Mereka menunjukkan bahwa scFv-phages terikat untuk memelihara lisozim putih telur, tetapi bukan lisozim manusia atau kalkun eggwhite lisozim. Kekhususan yang sangat tinggi menunjukkan bahwa scFv ditampilkan secara fungsional bentuk terlipat pada permukaan phage. Pengayaan phage jutaan kali yang membawa antigenbinding fragmen atas phage lain dicapai dengan menggunakan dua putaran pemurnian afinitas dengan mengintervensi amplifikasi dari phage yang ditahan.
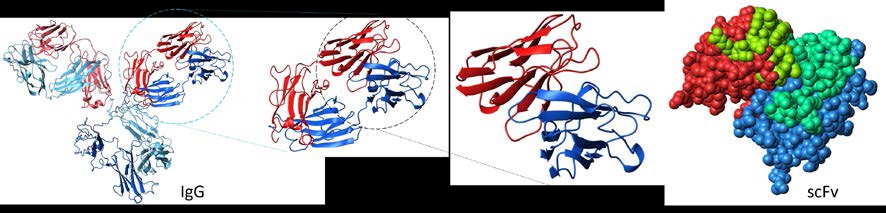
Gambar 7. Definisi fragmen antibodi. Struktur Immunoglobulin G (IgG) (kiri) dengan dua berat rantai biru dan biru muda dan dua rantai cahaya merah dan merah muda, dengan lokasi Fab dan scFv (89) ditunjukkan. Model pengisian ruang dari scFv (kanan) menunjukkan loop variabel berwarna hijau.
Pencapaian ini adalah titik awal dari revolusi farmasi: keampuhan yang terbukti menggunakan tampilan phage dari fragmen antibodi fungsional untuk pemilihan pengikat afinitas tinggi untuk antigen yang ditentukan. Teknologi sekarang memungkinkan untuk memotong imunisasi untuk generasi antibodi monoklonal dengan perancah manusia sepenuhnya, dan kemungkinan untuk ditampilkan perpustakaan kombinatorial antibodi manusia memfasilitasi pengembangan terapeutik antibodi. Selain itu, teknologi layar phage memungkinkan untuk memperoleh antibodi mengenali antige beracun, atau antigen yang akan membahayakan tuan rumah. Terinspirasi oleh fag-display pustaka peptida, Winter menguraikan beberapa masa depan arah dan aplikasi tampilan phage antibodi. Dia mengusulkan pembangunan dan skrining perpustakaan antibodi kombinatorial besar yang dibuat oleh kombinasi acak gen untuk domain variabel rantai berat dan ringan untuk ditampilkan pada phage berfilamen. Dia juga mengusulkan produksi dan penyaringan dari perpustakaan antibodi sepenuhnya sintetis pada phage.
Contoh lain dari tampilan phage fragmen antibodi, dalam hal ini sepanjang keseluruhan permukaan fag, dalam fusi dengan major coat protein VIII, dilaporkan oleh Richard Lerner dan rekan kerja pada tahun 1991.(90) Karya ini menampilkan fragmen antibodi yang lebih besar, yang disebut Fragmen Fab, diturunkan pada tikus terhadap tetanus toxin. Dua domain dari antibodi yang berat rantai ditampilkan pada fag dan dua domain dari rantai cahaya antibodi diekspresikan dalam bentuk yang disekresikan. Potongan-potongan dari kedua rantai itu membentuk kembali Fab di permukaan fag dan pengayaan 2.700 kali lipat selama fag non-spesifik dicapai dengan menggunakan biopanning; itu peneliti mengusulkan bahwa pengayaan yang lebih tinggi akan muncul dengan menggunakan putaran biopanning berurutan.
Juga pada tahun 1991, tampilan phage dari fragmen Fab heterodimeric dari tikus yang diimunisasi pada protein III dilaporkan oleh Winter dan rekan kerja.(91) Dua domain dari satu rantai antibodi (berat atau cahaya) diekspresikan dalam fusi dengan protein phage III dan dua domain dari antibodi yang berlawanan rantai (ringan atau berat, masing-masing) dalam bentuk yang disekresikan. Kedua orientasi ditemukan menghasilkan 15 Fab fungsional; kedua fragmen tersebut menyusun kembali Fab di permukaan filage filamen. Itu beralasan bahwa ini menyediakan format untuk menjelajahi rangkaian urutan yang jauh lebih luas dari yang dapat dicapai dalam perpustakaan tunggal (107) melalui keunggulan kombinatif dari coexpressing dua perpustakaan.(91) Studi lain dari tahun 1991 menampilkan fragmen Fab mouse yang diarahkan terhadap domain ekstraseluler reseptor HER2, dan menunjukkan bahwa itu bisa diperkaya juta lipat dari latar belakang phage yang tidak mengikat,(92) atau diperkaya relatif terhadap Fab termutasi fragmen dengan afinitas yang lebih rendah untuk antigen yang sama.
Tampilan phage dari perpustakaan antibodi
Sejumlah perpustakaan antibodi ditampilkan pada phage berfilamen dilaporkan selama tahun 1991. Pekerjaan ini dikejar di beberapa laboratorium secara paralel. Tampilan perpustakaan gabungan fragmen Fab pada protein permukaan fagsi III dilaporkan.(93) Perpustakaan ini berisi pecahan kecil fragmen Fab yang berasal dari tikus yang diimunisasi dengan toksin tetanus. Perpustakaan digunakan untuk memilih pengikat untuk toksin tetanus dari latar belakang phage tidak mengikat menggunakan biopanning.(93) Publikasi lain melaporkan Fab-library yang ditampilkan phage dari 10 juta varian urutan, yang berasal dari sumsum tulang manusia HIV-positif.(94) Perpustakaan ini digunakan untuk menurunkan fragmen afinitas tinggi terhadap glikoprotein gp120 dari virus.
Dua perpustakaan scFv dari manusia dilaporkan pada tahun 1991, ditampilkan pada protein III dari phage berfilamen.(95) Perpustakaan ini dibangun sebagai kombinasi dari bagian variabel dari rantai berat dan ringan IgM (27 juta anggota) atau bagian variabel dari rantai berat dan ringan IgG (160 juta anggota) yang berasal dari manusia yang tidak diimunisasi. Kemampuan untuk memperoleh antibodi afinitas tinggi dari pustaka-pustaka ini menggunakan empat putaran biopanning ditunjukkan dengan menggunakan antigen sebagai phox molekul kecil, serta lisozim protein dari albumin telur-putih kalkun dan bovin.
Perpustakaan awal antibodi
Pada tahun 1989, Winter dan rekan kerja melaporkan perpustakaan fragmen antibodi yang dapat ditangani di luar sel untuk seleksi pada vitro.(96) Reproduksi asli fragmen VH antibodi berasal dari tikus yang diimunisasi dengan lysozyme atau haemocyanine dan dikloning dalam format yang memungkinkan sekresi dari E. coli, sendiri atau dalam kombinasi dengan rantai VL (Vðž³ðž³). Antibodi dapat dipilih dari perpustakaan yang disekresikan menggunakan antigen teramobilisasi. Dalam contoh kedua dari tahun yang sama, Lerner dan rekan kerja menyatakan perpustakaan kombinasi besar fragmen Fab mewakili repertoar antibodi tikus dalam E. coli menggunakan vektor lambda. (97)(98)Skrining perpustakaan ini untuk anggota pengikatan antigen dilakukan dengan menggunakan uji pengangkatan plak. Teknologi yang digunakan dalam upaya sebelumnya untuk seleksi dan penyaringan perpustakaan antibodi segera ditinggalkan demi teknologi tampilan phage yang lebih efisien.(99-102)
Format tampilan lainnya
Layar phage adalah contoh pertama dari metode evolusi protein pengikat, yang menawarkan kopling fisik fenotipe (urutan protein) dan genotipe (urutan DNA). Beberapa metode tampilan pustaka lainnya yang memiliki fenotipe pasangan terhadap genotipe telah dikembangkan, misalnya, tampilan ribosom,(103)(104) tampilan bakteri (105) dan tampilan ragi.(106) Namun, tampilan phage tetap menjadi platform yang paling banyak digunakan untuk derivasi antibodi dan fragmen antibodi, dengan dampak medis yang sangat besar.
Aplikasi dan prospek
Evolusi yang diarahkan pada protein yang mengikat telah menjadi protokol yang sangat efisien untuk pengembangan antibodi terapeutik. Tampilan phage dari peptida dan antibodi digunakan untuk menurunkan varian dengan afinitas tinggi untuk target yang diberikan, dan untuk memberikan informasi berharga mengenai persyaratan urutan dan gaya mengemudi molekuler untuk afinitas dan spesifisitas dalam reaksi pengikatan, pelipatan protein dan stabilitas.
Sutradara evolusi untuk meningkatkan afinitas pengikatan sementara mempertahankan selektivitas tinggi antibodi telah digunakan dalam 25 tahun terakhir. Seperti "pematangan afinitas" prosedur telah memperoleh antibodi dengan afinitas untuk antigen mereka dalam kisaran 1012-1015 M-1 mewakili peningkatan seribu hingga juta lipatan atas antibodi yang dihasilkan dari imunisasi, dan kemudian dari perpustakaan menampilkan phage. Afinitas yang sangat tinggi memungkinkan penggunaannya sebagai agen terapeutik subkutan, yang diberikan sendiri, daripada membutuhkan jumlah yang lebih besar yang disuntikkan secara intravena di ruang dokter.
Antibodi terapeutik manusia sepenuhnya yang pertama diperoleh dengan menggunakan tampilan phage adalah Adalimumab (disetujui 2002). Adalimumab mengikat dengan afinitas yang sangat tinggi terhadap protein yang disekresikan TNF-, sitokin pro-inflamasi, dan digunakan dalam pengobatan rheumatoid arthritis, psoriasis danpenyakit radang usus, di antara kondisilainnya. Berbagai antibodi manusia telah dan akan diidentifikasi menggunakan teknologi tampilan phage dan dalam penggunaan klinis terhadap misalnya penyakit radang dan kanker.
DAFTAR PUSTAKA:
(1) Eigen M, Gardiner W .1984. Evolutionary molecular engineering based on RNA replication. Pure Appl. Chem. 56, 967-978.
(2) Guterman and Ladner.1991. EP0436597 B, published 17 Juli 1991, priority date 2 sep 1988.
(3) Chen KQ, Arnold FH .1991. Enzyme engineering for nonaqueous solvents: random mutagenesis to enhance activity of subtilisin E in polar organic media. Biotechnology (N Y). 9:1073-1077.
(4) Chen K, Arnold FH .1993. Tuning the activity of an enzyme for unusual environments: sequential random mutagenesis of subtilisin E for catalysis in dimethylformamide. Proc Natl Acad Sci U S A. 90:5618-5622.
(5) Arnold FH .1993. Engineering proteins for unusual environments. FASEB J 7:744-749.
(6) Stemmer WP .1994. Rapid evolution of a protein in vitro by DNA shuffling. Nature 370:389-391.
(7) Stemmer WP .1994. DNA shuffling by random fragmentation and reassembly: in vitro recombination for molecular evolution. Proc Natl Acad Sci U S A. 91:10747-10751.
(8) Clackson T, Hoogenboom HR, Bonnert TP, McCafferty J, Griffiths AD, Winter G .1991. Making antibody fragments using phage display libraries. Nature 352:624-628.
(9) Marks JD, Griffiths AD, Malmqvist M, Clackson TP, Bye JM, Winter G. .1992. By-passing immunization: Building high affinity human antibodies by chain shuffling. Nat. Biotech. 10, 779-783.
(10) Collet TA, Roben P, O’Kennedy R Barbas III, CF, Burton DR, Lerner RA .1992. A binary plasmid system for shuffling combinatorial antibody libraries. Proc Natl Acad Sci U S A. 89:10026-10030.
(11) Crameri A, Stemmer WP .1995. Combinatorial multiple cassette mutagenesis creates all the permutations of mutant and wild-type sequences. Biotechniques 18:194-196.
(12) Crameri A, Whitehorn EA, Tate E, Stemmer WP .1996. Improved green fluorescent protein by molecular evolution using DNA shuffling. Nat Biotechnol 14:315-319.
(13) Miyazaki K, Arnold FH .996. Exploring nonnatural evolutionary pathways by saturation mutagenesis: rapid improvement of protein function. J Mol Evol. 49:716-720.
(14) Crameri A, Dawes G, Rodriguez E Jr, Silver S, Stemmer WP .1997. Molecular evolution of an arsenate detoxification pathway by DNA shuffling. Nat Biotechnol. 15:436-438.
(15) Zhang JH, Dawes G, Stemmer WP .1997. Directed evolution of a fucosidase from a galactosidase by DNA shuffling and screening. Proc Natl Acad Sci U S A. 94:4504-4509.
(16) Moore JC, Jin HM, Kuchner O, Arnold FH .1997. Strategies for the in vitro evolution of protein function: enzyme evolution by random recombination of improved sequences. J Mol Biol. 272:336-347.
(17) Zhao H, Arnold FH .1997. Functional and nonfunctional mutations distinguished by random recombination of homologous genes. Proc Natl Acad Sci U S A. 94:7997-8000.
(18) Zhao H, Arnold FH .1997. Optimization of DNA shuffling for high fidelity recombination. Nucleic Acids Res. 25:1307-1308.
(19) Zhao H, Giver L, Shao Z, Affholter JA, Arnold FH .1998. Molecular evolution by staggered extension process (StEP) in vitro recombination. Nat Biotechnol 16:258-61.
(20 Crameri A, Raillard SA, Bermudez E, Stemmer WP .1998. DNA shuffling of a family of genes from diverse species accelerates directed evolution. Nature 391:288-291.
(21) Shao Z, Zhao H, Giver L, Arnold FH .1998. Random-priming in vitro recombination: an effective tool for directed evolution. Nucleic Acids Res 26:681-683.
(22) Miyazaki K, Arnold FH .1999. Exploring non-natural evolutionary pathways by saturation mutagenesis: rapid improvement of protein function. J Mol Evol 49:716-720.
(23) Christians FC, Scapozza L, Crameri A, Folkers G, Stemmer WP .1999. Directed evolution of thymidine kinase for AZT phosphorylation using DNA family shuffling. Nat Biotechnol 17:259-264.
(24) Ostermeier M, Sinn JH, Benkovic SJ .1999. A combinatorial approach to hybrid enzymes independent of DNA homology. Nat Bioctech 17:1205-1209.
(25) Sieber V, Martinez CA, Arnold FH .2001. Libraries of hybrid proteins from distantly related sequences. Nat biotechnol 19:456-460.
(26) Voigt CA, Martinez C, Wang ZG, Mayo SL, Arnold FH .2002. Protein building blocks preserved by recombination. Nat Struct Biol 9:553-558.
27. Rice GC, Goeddel DV, Cachianes G, Woronicz J, Chen EY, Williams SR, Leung DW .1992. Random PCR mutagenesis screening of secreted proteins by direct expression in mammalian cells. Proc Natl Acad Sci USA 89:5467-5471.
(28) Fu AY, Spence C, Scherer A, Arnold FH, Quake SR .1999. A microfabricated fluorescence-activated cell sorter. Nat Biotechnol 17:1109-1111.
(29) Tawfik DS, Griffiths AD .1998. Man-made cell-like compartments for molecular evolution. Nat Biotechnol 16:652-656.
(30) You L, Arnold FH .1994. Directed evolution of subtilisin E in Bacillus subtilis to enhance total activity in aqueous dimethylformamide. Protein Eng 1996;9:77–83.
(31) Moore JC, Arnold FH .1996. Directed evolution of a para-nitrobenzyl esterase for aqueous-organic solvents. Nat Biotechnol 14:458-467.
(32) Giver L, Gershenson A, Freskgard PO, Arnold FH .1998. Directed evolution of a thermostable esterase. Proc Natl Acad Sci U S A. 95:12809-12813.
(33) Zhao HM, Arnold FH .1999. Directed evolution coverts subtilisin E into a functional equivalent of thermitase. Protein Eng. 12:47-53.
(34) Wintrode PL, Miyasaki K, Arnold FH .2000. Cold adaption of a mesophilic subtilisin-like protease by laboratory evolution. J Biol Chem 275:31635-31640.
(35) Wintrode PL, Miyasaki K, Arnold FH .2001. Patterns of adaption of a mesophilic subtilisin-like protease by laboratory evolution. BBA Protein Struct Mol Evol 1549:1-8.
(36) Reetz MT, Carballeira JD, Vogel A.2001. Iterative saturation mutagenesis on the basis of B factors as a strategy for increasing protein thermostability. Angew Chem Int Ed 2006;45:7745–7751.
(37) Bloom JD, Wilke CO, Arnold FH, Adami C .2004. Stability and evolvability of function in model proteins. Biophys J 86:2758-2764.
(38) Bloom JD, Labthavikul ST, Otey CR, Arnold FH .2006. Protein stability promotes evolvability. Proc Natl Acad Sci USA 103:5869-5874.
(39) Drummond DA, Siberg JJ, Meyer MM, Wilke CO, Arnold FH .2005. On the conservative nature of intragenic recombination. Proc Natl Acad Sci USA 102:5380-5385.
(40) Bloom JD, Romero PA, Lu Z, Arnold FH .2007. Neutral drift ca alter promiscuous protein functions, potentially altering functional evolution. Biology Direct 2.
(41) Bershtein S, Segal M, Bekerman R, Tokuriki N, Tawfik D .2006. Robustness-epistasis link shapes the fitness landscape of a randomly drifting protein. Nature 444:929-932.
(42) Drummond DA, Bloom JD, Adami C, Wilke CO, Arnold FH .2005. Why highly expressed proteins evolve slowly. Proc Natl Acad Sci USA 102:14338-14343.
(43) Kazlauskas RJ .2005. Enhancing catalytic promiscuity for biocatalysis. Curr Opin Chem Biol. 9:195-201.
(44) Bornscheuer UT, Kazlauskas RJ .2004. Catalytic promiscuity in biocatalysis: using old enzymes to form new bonds and follow new pathways. Angew Chem Int Ed Engl 43:6032-6040.
(45) Arnold FH .2015. The nature of chemical innovation: new enzymes by evolution. QRB 48:404-410.
(46) Arnold FH .2018. Directed evolution: bringing new chemistry to life. Angew. Chem. Int. Ed. 57:4143-4148.
(47) Buller AR, Brinkmann-Chen S, Romney DK, Herger M, Murciano-Calles J, Arnold FH .2015. Directed evolution of the tryptophan synthase -subunit for stand-alone function recapitulates allosteric activation. Proc Natl Acad Sci U S A 112:14599-14604.
(48) Herger M, van Roye P, Romney DK, Brinkmann-Chen S, Buller AR, Arnold FH .2016. Synthesis of -Branched Tryptophan Analogues Using an Engineered Subunit of Tryptophan Synthase. J Am Chem Soc. 138:8388-8391.
(49) Murciano-Calles J, Romney DK, Brinkmann-Chen S, Buller AR, Arnold FH .2016. A Panel of TrpB Biocatalysts Derived from Tryptophan Synthase through the Transfer of Mutations that Mimic Allosteric Activation. Angew Chem Int Ed Engl 55:11577-11581.
(50) Boville CE, Romney DK, Almhjell PJ, Sieben M, Arnold FH .2018. Improved Synthesis of 4-Cyanotryptophan and Other Tryptophan Analogues in Aqueous Solvent Using Variants of TrpB from Thermotoga maritima. J Org Chem 83:7447-7452.
(51) Buller AR, van Roye P, Cahn JKB, Scheele RA, Herger M, Arnold FH .2018. Directed Evolution Mimics Allosteric Activation by Stepwise Tuning of the Conformational Ensemble. J Am Chem Soc 140:7256-7266.
(52) Coelho PS, Wang ZJ, Ener ME, Baril SA, Kannan A, Arnold FH, Brustad EM .2013. A serine-substituted P450 catalyzes highly efficient carbene transfer to olefins in vivo. Nat Chem Biol. 9:485-487.
(53) Coelho PS, Brustad EM, Kannan A, Arnold FH .2013. Olefin cyclopropanation via carbene transfer catalyzed by engineered cytochrome P450 enzymes. Science 339:307-310.
(54) Wang ZJ, Renata H, Peck NE, Farwell CC, Coelho PS, Arnold FH .2014. Improved cyclopropanation activity of histidine-ligated cytochrome P450 enables the enantioselective formal synthesis of levomilnacipran. Angew Chem Int Ed Engl. 53:6810-6813.
(55) Prier CK, Hyster TK, Farwell CC, Huang A, Arnold FH .2016. Asymmetric Enzymatic Synthesis of Allylic Amines: A Sigmatropic Rearrangement Strategy. Angew Chem Int Ed Engl. 55:4711-4715.
(56) Brandenberg OF, Fasan R, Arnold FH .2017. Exploiting and engineering hemoproteins for abiological carbine and nitrene transfer reactions. Curr Opin Biotechnol. 47:102-111.
(57) Lewis RD, Garcia-Borràs M, Chalkley MJ, Buller AR, Houk KN, Kan SBJ, Arnold FH .2018. Catalytic iron-carbene intermediate revealed in a cytochrome c carbene transferase. Proc Natl Acad Sci U S A 115:7308-7313.
(58) Prier CK, Zhang RK, Buller AR, Brinkmann-Chen S, Arnold FH .2017. Enantioselective, intermolecular benzylic C-H amination catalysed by an engineered iron-haem enzyme. Nat Chem. 9:629-634.
(59) Chen K, Huang X, Kan SBJ, Zhang RK, Arnold FH .2018. Enzymatic construction of highly strained carbocycles. Science 360:71-75.
(60) Schmidt-Dannert C, Umeno D, Arnold FH .2000. Molecular breeding of carotenoid biosynthetic pathways. Nat biotechnol 18:750-753.
(61) May O, Nguyen PT, Arnold FH .2000. Inverting enantioselectivity by directed evolution of hydantoinase for improved prodcutionof L-methionine. Nat biotechnol 18:317-320.
(62) Glieder A, Farinas ET, Arnold FH .2002. Laboratory evolution of a soluble, self-sufficient, highly active alkane hydroxylase. Nature Biotech 20:1135–1139.
(63) Bastian S, Liu X, Meyerowitz JT, Snow CD, Chen MMY, Arnold FH .2011. Engineered ketol-acid reductoisomerase and alcoholdehydrogenase enable anaerobic 2-methylpropan-1-ol production a ttheoretical yield in Escherichia coli. Metabolic Engineering 13 (2011) 345–352.
(64) Kan SB, Lewis RD, Chen K, Arnold FH .2016. Directed evolution of cytochrome c for carbon-silicon bond formation: Bringing silicon to life. Science 354:1048-1051.
(65) Kan SBJ, Huang X, Gumulya Y, Chen K, Arnold FH .2017. Genetically programmed chiral organoborane synthesis. Nature 552:132-136.
(66) McIntosh JA, Coelho PS, Farwell CC, Wang ZJ, Lewis JC, Brown TR, Arnold FH .2013. Enantioselective intramolecular C-H amination catalyzed by engineered cytochrome P450 enzymes in vitro and in vivo. Angew Chem Int Ed Engl 52:9309-9312.
(67) Matcham GW, Bowen ARS .1996. Biocatalysis for chiral indermediates: meeting commercial and technical challenges. Chimica Oggi (Chemistry Today) 14, 20-24.
(68) Reetz, M.T., Zonta, A., Schimossek, K., Liebeton, K. & Jaeger, K.-E. 1997. Creation of enantioselective biocatalysts for organic chemistry by in vitro evolution. Angew. Chem. Int. Ed. Engl. 36, 2830-2832.
(69) Liebeton K, Zonta A, Schimossek K, Nardini M, Lang D, Dijkstra BW, Reetz MT, Jaeger KE .2000. Directed evolution of an enantioselective lipase. Chem Biol. 7:709-718.
(70) Reetz, M.T., Becker, M.H., Klein, H.-W. & Stoëckigt, D.A. 1999. Method for high-throughput screening of enantioselective catalysts. Angew. Chem. Int. Ed. 38, 1758-1761.
22 (24)
(71) Reetz MT, Jaeger KE .2000. Enantioselective enzymes for organic synthesis, created by directed evolution Chemistry 6:407-412.
(72) Reetz MT .2000. Evolution in the test tube as a means to create enantioselective enzymes for use in organic synthesis. Sci Prog. 83(Pt2):157-172.
(73). Jaeger KE, Reetz MT .2000. Directed evolution of enantioselective enzymes for organic chemistry. Curr Opin Chem Biol 4:68-73.
(74) Fasan R, Crook NC, Peters MW, Meinhold P, Buelter T, Landwehr M, Cirino PC, Arnold FH .2011. Improved product-per-glucose yields in P450-dependent propane biotransformations using engineered Escherichia coli. Biotechnol Bioeng. 108:500-10.
(75) Fischbach MA, Lai JR, Roche ED, Walsh CT, Liu DR .2007. Directed evolution can rapidly improve the activity of chimeric assembly-line enzymes. Proc Natl Acad Sci USA 104:11951.
(76) Bajaj P, Sreenilayam G, Tyagi V, Fasan R .2016. Gram-Scale Synthesis of Chiral Cyclopropane-Containing Drugs and Drug Precursors with Engineered Myoglobin Catalysts Featuring Complementary Stereoselectivity. Angew Chem Int Ed Engl. 55:16110-16114.
(77) Hernandez KE, Renata H, Lewis RD, Jennifer Kan SB, Zhang C, Forte J, Rozzell D, McIntosh JA, Arnold FH .2016. Highly Stereoselective Biocatalytic Synthesis of Key Cyclopropane Intermediate to Ticagrelor. ACS Catal. 6:7810-7813.
(78) Jäckel C, Kast P, Hilvert D .2008. Protein design by directed evolution. Annu Rev Biophys 37:153-173.
(79) Althoff EA, Wamg L, Jiang L, Giger L, Lassila JK, Wang Z, Smith M, Hari S, Kasp P, Hersclag D, Hilvert D, Baker D. 2012. Robust design and optimization of retroaldol enzymes. Protein Sci 21:717-726.
(80) Obexer R, Godina A, Garrabau X, Mitti PR, Baker D, Griffiths AD, Hilvert D .2017. Emergence of a catalytic tetrad during evolution of a highly active artificial aldolase. Nat Chem 9:50-56.
(81) Smith GP .1985. Filamentous fusion phage: novel expression vectors that display cloned antigens on the virion surface. Science 228:1315-1317.
(82) Parmley SF, Smith GP .1988. Antibody-selectable filamentous fd phage vectors: affinity purification of target genes. Gene 73:305-318.
(83) de la Cruz VF, Lal AA, McCutchan TF .1988. Immunogenicity and epitope mapping of foreign sequences via genetically engineered filamentous phage. J Biol Chem 263:4318-4322.
(84) Greenwood J, Willis AE, Perham RN .1991. Multiple display of foreign peptides on a filamentous bacteriophage. Peptides from Plasmodium falciparum circumsporozoite protein as antigens. J Mol Biol. 1991, 220:821-827.
(85) DevlinJJ, Panganiban LC, Devlin PE .1990. Random peptide libraries: a source of specific protein-binding molecules. Science 249:404-406.
(86) Scott JK, Smith GP .1990. Searching for peptide ligands with an epitope library. Science 249:386-390.
(87) Cwirla SE, Peters EA, Barrett RW, Dower WJ .1990. Peptides on phage: a vast library of peptides for identifying ligands. Proc Natl Acad Sci USA 87:6378-6382.
(88) McCafferty J, Griffiths AD, Winter G, Chiswell DJ .1990. Phage antibodies: filamentous phage displaying antibody variable domains. Nature 348:552-554.
(89) Huston JS, Levinson D, Mudgett-Hunter M, Tai MS, Novotný J, Margolies MN, Ridge RJ, Bruccoleri RE, Haber E, Crea R .1988. Protein engineering of antibody binding sites: recovery of specific activity in an anti-digoxin single-chain Fv analogue produced in Escherichia coli. Proc Natl Acad Sci U S A 85:5879-5883.
23 (24)
(90) Kang AS, Barbas CF, Janda KD, Benkovic SJ, Lerner RA .1991. Linkage of recognition and replication functions by assembling combinatorial antibody Fab libraries along phage surfaces. Proc Natl Acad Sci U S A 88:4363-4366.
(91) Hoogenboom HR, Griffiths AD, Johnson KS, Chiswell DJ, Hudson P, Winter G .1991. Multi-subunit proteins on the surface of filamentous phage: methodologies for diplaying antibody (Fab heavy and light chains. Nucl. Acid. Res. 19:4133-4137.
(92) Garrard LJ, Yang M, O'Connell MP, Kelley RF, Henner DJ .1991. Fab assembly and enrichment in a monovalent phage display system. Biotechnology 9:1373-1377.
(93) Barbas CF, Kang AS, Lerner RA, Benkovic SJ .1991. Assembly of combinatorial antibody libraries on phage surfaces: the Gene III site. Proc Natl Acad Sci U S A. 88:7978-7982.
(94) Burton DR, Barbas CF 3rd, Persson MA, Koenig S, Chanock RM, Lerner RA .1991. A large array of human monoclonal antibodies to type 1 human immunodeficiency virus from combinatorial libraries of asymptomatic seropositive individuals. Proc Natl Acad Sci U S A. 88:10134-10137.
(95) Marks JD, Hoogenboom HR, Bonnert TP, McCafferty J, Griffiths AD, Winter G. .1991. By-passing immunization: Human antibodies from V-gene libraries displayed on phage. J Mol Biol 222:581-597.
(96) Ward ES, Güssow D, Griffiths AD, Jones PT, Winter G .1989. Binding activities of a repertoire of single immunoglobulin variable domains secreted from Escherichia coli. Nature 341:544-546.
(97) Huse WD, Sastry L, Iverson SA, Kang AS, Alting MM, Burton DR, Benkovic SJ, Lerner RA 1989. Generation of a large combinatorial library of the immunoglobulin repertoire in phage lambda. Science 246:1275-1281.
(98) Smith GP .1988. Filamentous phage assembly: morphogenetically defective mutants that do no ill the host. Virology 157:156-165.
(99) Hoogenboom HR, Winter G .1992. By-passing immunisation. Human antibodies from synthetic repertoires of germline VH gene segments rearranged in vitro. J Mol Biol 227:381-388.
(100) Bass S, Greene R, Wells JA .1990. Hormone phage: an enrichment method for variant proteins with altered binding properties. Proteins 8:309-314.
(101) Lowman HB, Bass SH, Simpson N, Wells JA .1991. Selecting high-affinity binding proteins by monovalent phage display. Biochemistry 30:10832-10838.
(102) Lowman HB, Wells JA .1993. Affinity maturation of human growth hormone by monovalent phage display. J Mol Biol 5:564-568.
(103) Mattheakis L, Bhatt R, Dower W .1994. An in vitro polysome display system for identifying ligands from very large peptide libraries. 91:9022-9026.
(104) Hanes J, Plückthun A .1997. In viro selelction and evolution of functional proteins by using ribosome display. 94:4937-4942.
(105) Georgiou G, Staphopolous C, Daugherty P, Nayak AR, Iverson BL, Curtiss R 3rd .1997. Display of heterologous proteins on the surface of microorganisms: rom the screening if combinatorial libraries to live recombinant vaccines. Nature Bioctech. 15:29-34.
(106) Boder ET, Wittrup KD .1997. Yeast surface display for screening combinatorial polypeptide libraries. Nature Biotech. 15:553-557.
(107) Malmborg AC, Borrebaeck CA .1993. BIAcore as a tool in antibody engineering. J Immunol Methods 183:7-13.






Komentar berhasil disembunyikan.